放射光イメージング
ソフトウェア(SAKASなど)
データ・カルテシステムSAKAS(SAga light source data KArte System)
1.はじめに
当センターでは、放射光を利用したマイクロCTや位相イメージングにより取得した画像データについて、
- サンプルの名前や状態
- X線カメラの種類、露光条件
- ビームラインの状況
等の計測条件に加えて、フィルター関数など再構成の画像処理条件に至る全ての条件を統合的に取り扱い、保存できるデータ・カルテシステム(SAga ls KArte System:SAKAS、サーカス)の構築を進めています。SAKASでは、これまで計測制御ソフトや処理ソフトで個別に保存していた条件を、テキスト形式(windowsのiniファイルと同じ構成)で1個のカルテ(Tag)ファイルとして管理するため、どのような処理がどのような条件で行われたか容易に記録・確認することができます。
2.SAKASの仕様
各処理は、1個の「tagファイル」(データファイル名+’.tag’を推奨)に順に記録する。Tagファイルのフォーマットは、Windowsのiniファイルに準拠し、
[セクション]
パラメータ=***
;コメント
とする。計測における主なセクションはSample、BL_Cond、Imager、Methodとする。記録ルールは
- l大文字・小文字の区別はしない。
- lパラメータの順番は問わない。
- l独自のセクション、及びパラメータがあっても良い。
- l独自のパラメータは、処理側で任意に設定できる。
とする。ソフトウェア処理(Proc2以降)は処理を行う毎に最低限下記情報を追記していく。その他、処理で使用したパラメータ等(形式はフリー)も記載する。
- Method:string:処理の内容(背景除算、再構成など)
- File_Name:string:生成されたファイル名(フルパス標記)。
- Width:integer:上記データの横画素数
- Height:integer:上記データの縦画素数
- Format:integer:上記データのフォーマット
- 0: byte (8 bit 符号無し)、1: WORD (16 bit 符号無し)、 2: 実数単精度 (single)、 3: 実数倍制度(real)
- ST:連番スタート番号
- END:連番終了番号。
3.SAKASのソフトウェア群
SAKAS対応のソフトウェアは以下で構成しています。いずれもGithubでソースコードを含め公開中です。
※簡単な取扱説明書はこちら20211028_取説.docx
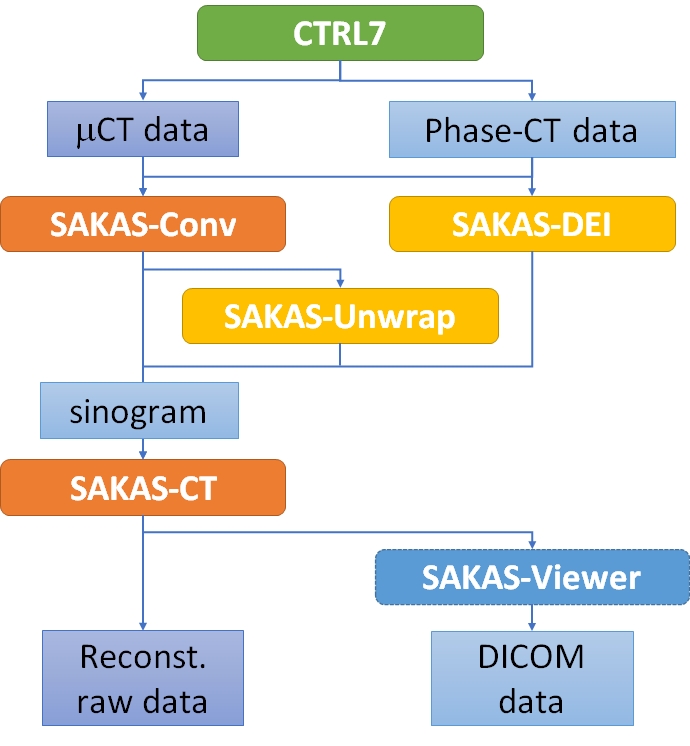 SAKASに対応したソフトウェア群
SAKASに対応したソフトウェア群
4.リングアーチファクト除去
サイノグラムにおける縦方向のハイパスフィルターと横方向のローパスフィルターの組合せにより、縦ストライプ、すなわちリングアーチファクトを低減するフィルター[1]を組み込んでいます。単純なフーリエ変換から構成されるため、パラメータの設定が単純で、高速かつ汎用性が高いことが特徴で、本機能により、検出器の感度ムラや入射X線強度の揺らぎに起因したリングアーチファクトを大幅に低減することが可能になります。
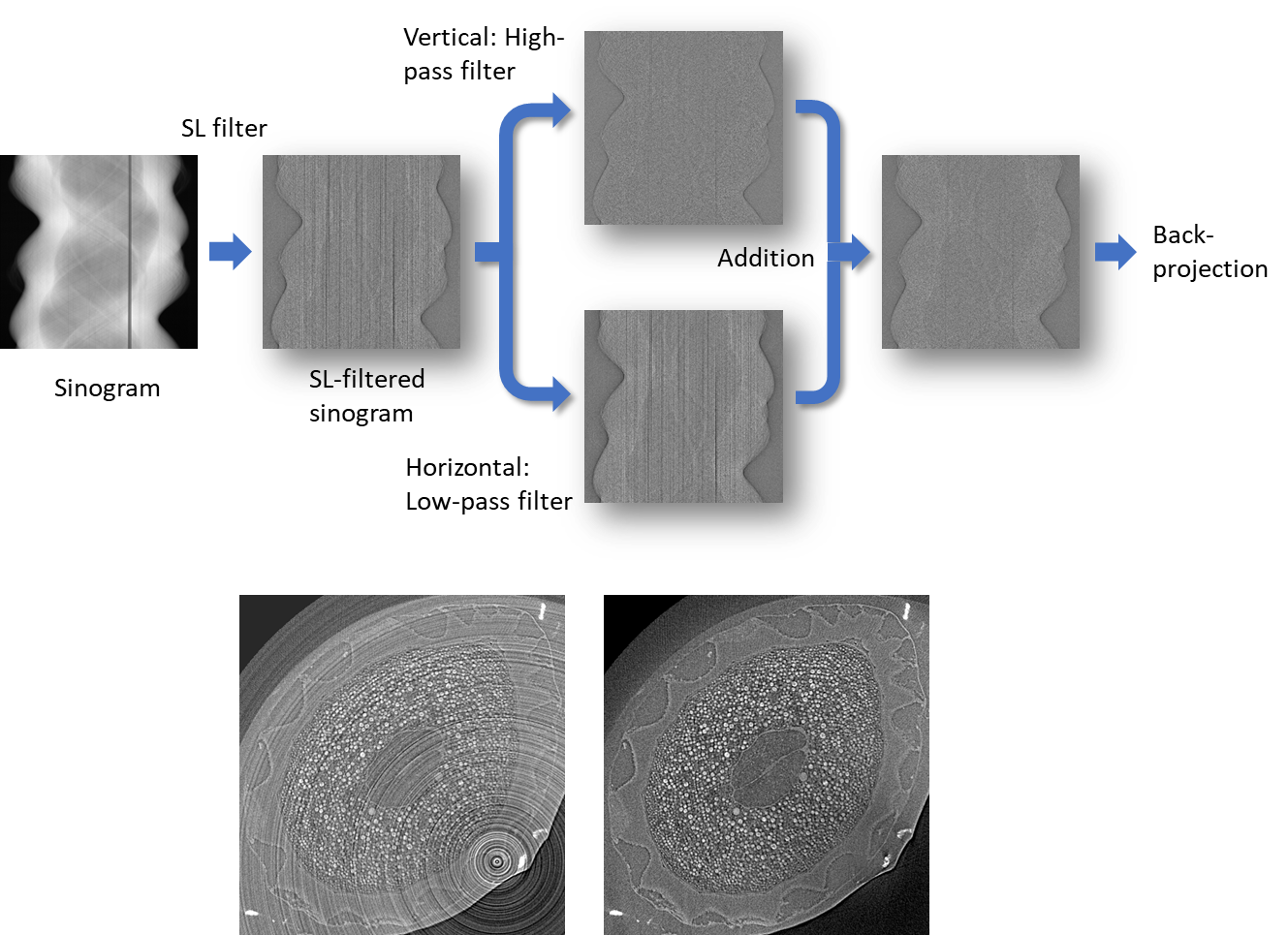 リングアーチファクト除去の概要とその例
リングアーチファクト除去の概要とその例
[1] A. Yoneyama, R. Baba, M. Kawamoto, T. T. LWIN, Method of ring artifact reduction in X-ray CT using 2-directional Fourier transform filter, ECR 2022, C-13289,
https://dx.doi.org/10.26044/ecr2022/C-13289
制御ソフト CTRL7
https://github.com/SAGALS-IMG/CTRL07
SAKASに対応した制御ソフトで制御対象機器は
- ツジ電子製 モーターコントローラ(PM16C、新制御コマンド)
- ツジ電子製 シャッターコントローラ(STR5C-01 )
- Andor製 sCMOSカメラ(Zyla)
- 温度コントローラ
です。
可能な計測法は
- 吸収CT
- 位相CT(縞走査法、アナライザー結晶スキャン法)
で、連続及びステップスキャンが選択可能です。
PDFマニュアル
SAKAS Conv
https://github.com/SAGALS-IMG/SAKAS-CONV
SAKASに対応したデータ変換ソフトです。
CTRL7で取得した画像データについて
- 背景画像による除算
- Sinogramの作成
を行います。
SAKAS CT
https://github.com/SAGALS-IMG/SAKAS-CT
SAKASに対応した断面像再構成ソフトです。
SAKAS Convで作成したSinogramについて
- SL関数などのコンボリューション(フーリエ面)
- リングアーチファクト除去処理[1]
- GPU或いはCPUを用いた逆投影
を行います。リングアーチファクト除去は上記に記載したとおりです。なお、本ソフトによる画像の保存形式は
ヘッダー:なし
フォーマット:64ビット浮動小数点(Double)
となります。
GPUはnVIDIAのCUDAを使用しています。GPUを使用して再構成計算を行うためには、CUDA 10.1のライブラリーをインストールしておく必要があります。ライブラリーはnVIDIAのHPから入手してください。
※本CT再構成はDEIの再構成(フィルター関数のコンボリューション前に、ステップ関数をコンボリューション)にも対応しています。
この機能は「Conv」タブの「Integration」をチェックすると機能します。通常のFBPでは、このチェックを外しておきます。
※本ソフトでは、ラインアーチファクトを制限するために、フィルター関数をコンボリューションした後に「Conv」タブのMin&Maxに入力された値で上限と下限を制限します。過度な制限は分解能の低下等を招きますので、データに合わせて最適な値を設定してください。
[1] https://epos.myesr.org/poster/esr/ecr2022/C-13289/References
SAKAS Viewer
https://github.com/SAGALS-IMG/SAKAS-VIEWER
SAKASに対応した画像ビューワソフトです。
元データから再構成したデータまで、SAKASで処理したデータであれば、Tagファイルを選択するだけで表示可能です。また、DICOMデータの書き出し機能も備えています(UIDにも対応)。さらに、画像の回転やシフトなどの基本的な機能に加えて、Medianフィルター、n値化、FFTを用いた周波数フィルター処理、2画像のマッチングやセグメンテーションなどの機能もあります。
SAKAS Viewerの主な機能
| 機能 | 特徴 | 機能 | 特徴 |
| B&C | 自動・手動で設定可能 | フーリエ変換 | ローパス、ハイパス、バンドバス |
| LUT | 擬似カラー化 | 統計処理 | 平均、標準偏差、ヒストグラム |
| 画像変形 | 回転、シフト、加算、Binning、Crop | DICOM書き出し | |
| フィルター | Median、n値化、Smoothing、エッジ強調 | マッチング | 2画像のマッチング |
以下DCMファイルをExportする簡易マニュアルです。
- SAKAS-Viewerを立ち上げる。
- ディレクトリーアイコンをクリックして新たに開いた「ファイルの選択」でTagファイルを選択
- 同ディレクトリー内のTagファイル一覧が表示されるので、開きたいデータのTagファイルを選択
- ProcでProc3を選択
- Img Openをクリックして、同データのProc3によって生成された画像ファイル(吸収CTの場合は再構成像)を開く
- 新しく開いた画像ビューアーでDICOMタブを選択
- 断面像内において赤い四角で切り出す領域を設定(ROI Onlyチェック時)
- Export Img No.でDICOM化する画像のスライス番号を指定
- HUの比例係数(HUD=□と表示されている□)を入力する。一般にLine profileから最大値を読み取り、最大値X比例係数が1000程度になるように、比例係数を決定する。
- 「DICOM Editor」でUIDなどが空欄でないことを確認。空欄の場合は、「Load DCM Header」ボタンを押し、SAKAS-Viewerと同じディレクトリーに保存されている「DCM.ini」を選択して、読み込む。次に「Import from SAKAS」ボタンを押して、Tagファイルを選択して、Tagファイルの情報を再度読み込む。
- 「Export」ボタンを押して、DCMデータをExportする。新しくDCMディレクトリーが作成され、各スライスのDCMデータが保存される。
※Alizaなどのボリュームレンダリングソフトで上記により生成したDCMファイルが読み込めない場合は、ヘッダーが正しくImportできていない可能性がある。この場合は、上記10を再度試してみる。
SAKAS DEI
https://github.com/SAGALS-IMG/SAKAS-DEI
SAKASに対応したDEIデータ処理ソフトです。
連続回転に対応し、任意点ノデータを利用してSinoguramを作成します。
SAKAS Unwrap
https://github.com/SAGALS-IMG/SAKAS-Unwrap
SAKASに対応した位相アンラップ処理ソフトです。
2πでラップされたデータについて、独自の距離計算方法により、2πを復元(アンラップ処理)します。
Image J (Fiji)について
SAKAS Viewerでサポートしていない高度な画像処理や統計解析等はImage Jを利用して実施することが可能です。以下簡単にImage Jの利用法について説明します。
Image Jのダウンロード先
https://imagej.net/software/fiji/downloads
(FijiはImage J+有用なPluginのセットです。)
画像の読み込み
- 読み込む再構成像のTagファイルを開きます。
- [Proc3]のWidthを確認します。Kenvy2で取得したデータでROIを設定していなければ1024 or 2058。Zyla 5.5HFで取得したデータでROIを設定していなければ2560 or 1280になります。
- Image Jを立ち上げます。
- メニューの「File」「Import」「Raw」をクリックします。
- 読み込みたいファイル(複数あればどれか1つ)を選択し、「開く」をクリックします。
- 新たに開いたWindowで以下を入力して「OK」ボタンを押す
- Image type:64-bit real
- Width:上記2で調べた値
- Height:Widthと同じ値
- Offset to first image:0
- Number of images:1
- Gap between images:0
- 「Little-endian byte order」及び「 Open all files in folder」をチェック
- ホルダー内の全ての再構成画像を読み込み(サイズと枚数によっては数分かかります。また、搭載メモリが少ないと読み込めない場合があります。)、表示します。
コントラストの調整
「Ctrl」「Alt」「Shift」キーを同時に押すと、「B&C」Windowが開き、「Auto」を押せば自動でコントラストを調整します。
多方向からのスライス画像
- 「Image」「Stacks」「Reslice [/]」をクリックします。
- 「Reslice」Windowで切り出す方向(Top, Left, Bottom, Right)を選択し、「OK」を押すと、選択した方向からのスライス像が表示されます。
注意
Image Jでは読み込む画像サイズだけPCのメモリを消費します。例えば、1024x1024画素からなる1000枚の再構成像の場合は約4G byte、更に別方向からのスライス画像を作ると計8G byteにもなります。このため、少なくともメモリを16G byte以上搭載したPCでの作業を推奨致します。なお、搭載メモリを超えて画像処理を続けた場合、HDDやSSDへのスワップを行い、極端に処理速度が低下し、場合によってはOSがハングします。
3次元ボリュームレンダリングについて
SAKAS ViewerのDICOM変換機能を利用して出力したCTデータは、下記に一例を示した有償および無償の3次元ボリュームレンダリングソフトウェア等を利用することで、3次元表示を行うことができます。なお、実際の利用に関しては、各ソフトウェアの説明書をご参照ください。
有償ソフトウェア
- ZioCube(Windows)
https://www.zio.co.jp/ziocube/ - OsiriX(Mac)
https://www.osirix-viewer.com/ - Aliza(Windows, Mac, Linux)
https://www.aliza-dicom-viewer.com/home
無償ソフトウェア
- 3D Slice(Windows, Mac, Linux)
https://slicer.readthedocs.io/en/latest/index.html - Para View(Windows)
https://www.paraview.org/ - MITK(Windows, Mac, Linux)
https://www.mitk.org/wiki/The_Medical_Imaging_Interaction_Toolkit_(MITK) - Horos(Mac)
https://horosproject.org/ - Seg3D(Windows)
https://www.sci.utah.edu/cibc-software/seg3d.html